Sponsored Links
生存時間分析は、イベントが起きるまでの時間とイベントとの間の関係に焦点を当てる分析方法です。こちらのページ [1] や、こちらのページ [2] が参考になります。生存時間分析では、Kaplan-Meier法を用いて生存曲線を描き、2群の生存曲線の比較としてLog-rank検定、イベントの発生を目的変数とした多重比較検定としてCox比例ハザード回帰分析などが行われます。
[1] EM Alliance – 第7回EMA-JC 解説 カプランマイヤー生存曲線 [2] saltcooky – Qiita – 生存時間分析の色々なアルゴリズムをまとめてみましたSponsored Links
データの準備:
survivalというパッケージに入っているkidneyというデータを使います。
> library(survival) # パッケージsurvivalを読み込みます
> head(kidney) # kidneyデータの先頭部分を表示
id time status age sex disease frail
1 1 8 1 28 1 Other 2.3
2 1 16 1 28 1 Other 2.3
3 2 23 1 48 2 GN 1.9
4 2 13 0 48 2 GN 1.9
5 3 22 1 32 1 Other 1.2
6 3 28 1 32 1 Other 1.2このデータは、ポータブル腎臓透析カテーテルを用いる際の感染発生までの時間を調べたものです。
time:イベント発生までの時間
status:イベント発生(1:感染発生、0:感染以外でカテーテル抜去)
age:年齢
sex:性別(1:男性、2:女性)
disease:GN:糸球体腎炎、AN:何の略か不明、PKD:多発性嚢胞腎、other:その他
frail:フレイルの推定
Kaplan-Meier法によるイベント発生までの時間の推定
生存時間分析は、一定期間、継続して観察を行うので、ターゲットとするイベントが生じる以外にも、何らかの要因で観察を打ち切らないといけない状況が出てきます。まず、パッケージsurvivalの中にあるSurvという関数で打ち切りを設定します。
> kidney.Surv <- Surv(kidney$time,kidney$status) # パッケージsurvivalの中にあるSurvという関数を使い、打ち切り情報を付加
> kidney.Surv # 数値は感染発生までの時間を示し、+が付加されている数値は感染以外の理由でで打ち切り
[1] 8 16 23 13+ 22 28 447 318 30 12 24 245 7 9 511 30
[17] 53 196 15 154 7 333 141 8+ 96 38 149+ 70+ 536 25+ 17 4+
[33] 185 177 292 114 22+ 159+ 15 108+ 152 562 402 24+ 13 66 39 46+
[49] 12 40 113+ 201 132 156 34 30 2 25 130 26 27 58 5+ 43
[65] 152 30 190 5+ 119 8 54+ 16+ 6+ 78 63 8+
> fit.disease <- survfit(Surv(time, status) ~ disease, data=kidney) # Kaplan-Meier法により疾患で層別化された感染までの時間を算出
> fit.disease
Call: survfit(formula = kidney.Surv ~ kidney$disease) # 疾患別の例数、イベント発生数、感染までの時間(中央値、95%CI)
n events median 0.95LCL 0.95UCL
kidney$disease=Other 26 20 141 30 318
kidney$disease=GN 18 14 30 25 NA
kidney$disease=AN 24 18 48 38 NA
kidney$disease=PKD 8 6 115 63 NA疾患別と同じように、性別で層別化された感染までの時間を算出してみます。
> fit.sex <- survfit(Surv(time, status) ~ sex, data=kidney) # Kaplan-Meier法により性別で層別化された感染までの時間を算出
> fit.sex
Call: survfit(formula = kidney.Surv ~ kidney$sex) # 性別ごとの例数、イベント発生数、感染までの時間(中央値、95%CI)
n events median 0.95LCL 0.95UCL
kidney$sex=1 20 18 22 12 63
kidney$sex=2 56 40 130 66 190Kaplan-Meier曲線の描画
上で作成した疾患により層別化されたデータを元に、Kaplan-Meier曲線を描画してみます。
> plot(fit.disease, main="Kaplan-Meier estimate", xlab="time", ylab="survival function", col=c("red", "blue", "green","pink"))
> legend("topright", legend=levels(kidney$disease), col=c("red","blue","green","pink"), lty=1)
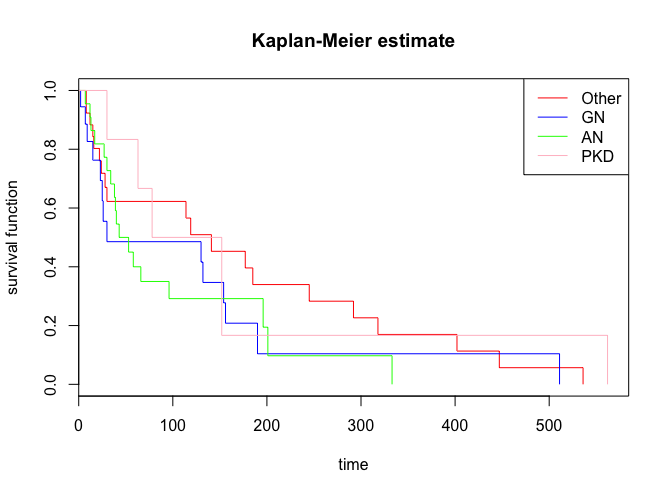
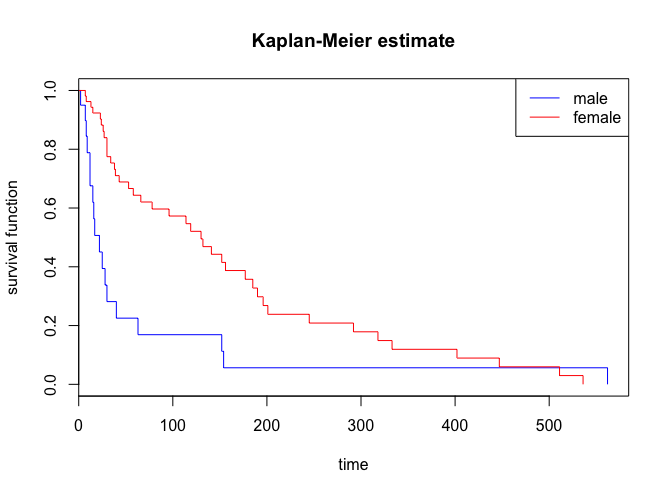
同様に、性別により層別化されたデータを元に、Kaplan-Meier曲線を描画してみます。
> plot(fit.sex, main="Kaplan-Meier estimate", xlab="time", ylab="survival function", col=c("blue", "red"))
> legend("topright", legend=c("male","female"), col=c("blue","red"), lty=1)
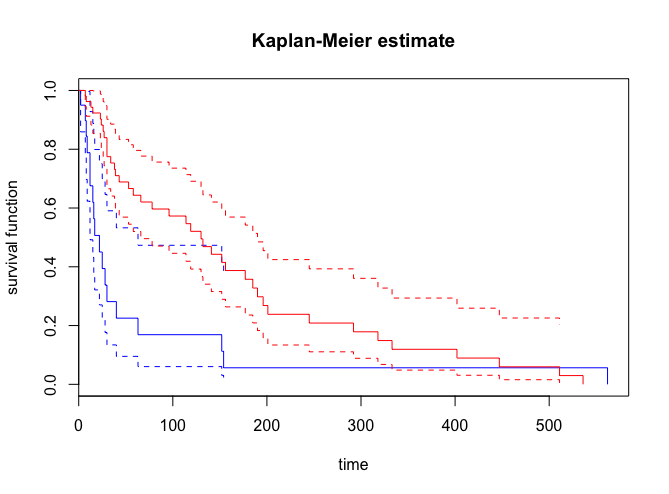
ちなみに、plotのオプションで、conf.int=TRUEを加えると95%信頼区間を描画できます。
> plot(fit.sex, main="Kaplan-Meier estimate", xlab="time", ylab="survival function", col=c("blue", "red"), conf.int=TRUE)
Log-rank検定
Log-rank検定は、層化されたKaplan-Meier曲線に差があるかどうかを調べる検定です。性別で層別化したKaplan-Meier曲線の差を検定してみます。[1]
> survdiff(Surv(time, status)~sex, data=kidney) # パッケージsurvivalの中のsurvdiff関数を使います。
Call:
survdiff(formula = Surv(time, status) ~ sex, data = kidney)
N Observed Expected (O-E)^2/E (O-E)^2/V
sex=1 20 18 10.2 5.99 8.31
sex=2 56 40 47.8 1.28 8.31
Chisq= 8.3 on 1 degrees of freedom, p= 0.004 次に疾患で層別化したKaplan-Meier曲線の差を検定してみます。
> survdiff(Surv(time, status)~disease, data=kidney)
Call:
survdiff(formula = Surv(time, status) ~ disease, data = kidney)
N Observed Expected (O-E)^2/E (O-E)^2/V
disease=Other 26 20 23.20 0.442 0.779
disease=GN 18 14 11.62 0.488 0.631
disease=AN 24 18 14.70 0.740 1.070
disease=PKD 8 6 8.48 0.724 0.989
Chisq= 2.7 on 3 degrees of freedom, p= 0.4 Lon-rank検定では疾患でのイベント発生に差は認められません。
[1] 比例ハザードモデルはとってもtricky!Cox比例ハザード回帰分析
Cox比例ハザード回帰分析は、共変量を考慮して生存曲線を比較する方法で、多変量解析のひとつです。時間を考慮したロジスティック回帰分析のようなものですね。ハザードというのはアウトカムが発生するリスクという意味で、比例ハザードは、そのリスクが時間の経過によらず一定ということだそうです。リスクが時間の経過によらず一定かどうかを簡単に見分ける方法として2つのKapran-Meier曲線が交差していたら、比例ハザードが成立しないと予想されるそうです [1]。
パッケージsurvivalの中にあるcoxph関数によりCox比例ハザードモデルのパラメータを推定できます。コックス比例ハザードモデルのパラメータの推定方法としては、直接法、Breslowの近似法、Efronの近似法などがあるそうです。coxph関数では、何も指定しないとEfronの近似法が用いられるようですが、method=””でefron、breslow、exactが指定でき、exactで直接法も指定できます [2]。
> kidney.cox <- coxph(Surv(time, status)~sex+disease, data = kidney) # sex+diseaseで説明変数に性別と疾患を指定
> summary(kidney.cox) # exp(coef)がハザード比ですね
Call:
coxph(formula = Surv(time, status) ~ sex + disease, data = kidney)
n= 76, number of events= 58
coef exp(coef) se(coef) z Pr(>|z|)
sex -1.4774 0.2282 0.3569 -4.140 3.48e-05 ***
diseaseGN 0.1392 1.1494 0.3635 0.383 0.7017
diseaseAN 0.4132 1.5116 0.3360 1.230 0.2188
diseasePKD -1.3671 0.2549 0.5889 -2.321 0.0203 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
exp(coef) exp(-coef) lower .95 upper .95
sex 0.2282 4.3815 0.11339 0.4594
diseaseGN 1.1494 0.8700 0.56368 2.3437
diseaseAN 1.5116 0.6616 0.78245 2.9202
diseasePKD 0.2549 3.9238 0.08035 0.8084
Concordance= 0.696 (se = 0.039 )
Rsquare= 0.206 (max possible= 0.993 )
Likelihood ratio test= 17.56 on 4 df, p=0.002
Wald test = 19.77 on 4 df, p=6e-04
Score (logrank) test = 19.97 on 4 df, p=5e-04method=”exact”を指定して直接法で計算してみます。
> kidney.cox.exact <- coxph(Surv(time, status)~sex+disease, method = "exact", data = kidney)
> summary(kidney.cox.exact)
Call:
coxph(formula = Surv(time, status) ~ sex + disease, data = kidney,
method = "exact")
n= 76, number of events= 58
coef exp(coef) se(coef) z Pr(>|z|)
sex -1.4972 0.2238 0.3627 -4.128 3.65e-05 ***
diseaseGN 0.1462 1.1574 0.3655 0.400 0.6891
diseaseAN 0.4257 1.5306 0.3381 1.259 0.2080
diseasePKD -1.3877 0.2496 0.5924 -2.343 0.0191 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
exp(coef) exp(-coef) lower .95 upper .95
sex 0.2238 4.4692 0.10992 0.4555
diseaseGN 1.1574 0.8640 0.56546 2.3691
diseaseAN 1.5306 0.6533 0.78900 2.9693
diseasePKD 0.2496 4.0057 0.07818 0.7972
Concordance= 0.696 (se = 0.039 )
Rsquare= 0.208 (max possible= 0.992 )
Likelihood ratio test= 17.73 on 4 df, p=0.001
Wald test = 19.64 on 4 df, p=6e-04
Score (logrank) test = 19.92 on 4 df, p=5e-04何も指定しないEfronの近似法より、method=”exact”と指定した方がハザード比が若干有意になっています。
coxph関数では、尤度比の検定(Likelihood ratio test)、ワルド検定(Wald test)、スコア検定(Score (logrank) test)が行われており、これらは説明変数が正規分布に従うことを仮定した検定になっています [2]。
coxphにより作られたモデルを当てはめて生存時間を推定するには、survfit関数を使います。
> kidney.cox.fit <- survfit(kidney.cox)
> summary(kidney.cox.fit)
Call: survfit(formula = kidney.cox)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
2 76 1 0.99018 0.00985 0.971059 1.000
7 71 2 0.96856 0.01831 0.933335 1.000
8 69 2 0.94641 0.02422 0.900102 0.995
9 65 1 0.93499 0.02685 0.883820 0.989
12 64 2 0.91106 0.03177 0.850869 0.976
13 62 1 0.89844 0.03411 0.834007 0.968
15 60 2 0.87273 0.03844 0.800550 0.951
16 58 1 0.85959 0.04046 0.783848 0.943
17 56 1 0.84591 0.04246 0.766654 0.933
22 55 1 0.83144 0.04446 0.748706 0.923
23 53 1 0.81625 0.04646 0.730084 0.913
24 52 1 0.80113 0.04830 0.711843 0.902
25 50 1 0.78592 0.05004 0.693710 0.890
26 48 1 0.76976 0.05184 0.674573 0.878
27 47 1 0.75369 0.05350 0.655802 0.866
28 46 1 0.73761 0.05503 0.637273 0.854
30 45 4 0.67000 0.06032 0.561621 0.799
34 41 1 0.65287 0.06137 0.543011 0.785
38 40 1 0.63574 0.06232 0.524602 0.770
39 39 1 0.61861 0.06317 0.506390 0.756
40 38 1 0.60147 0.06392 0.488373 0.741
43 37 1 0.58250 0.06463 0.468646 0.724
53 35 1 0.56288 0.06531 0.448382 0.707
58 33 1 0.54279 0.06594 0.427786 0.689
63 32 1 0.52270 0.06642 0.407467 0.671
66 31 1 0.50281 0.06677 0.387591 0.652
78 29 1 0.48209 0.06708 0.367010 0.633
96 28 1 0.46208 0.06717 0.347515 0.614
114 25 1 0.44051 0.06735 0.326443 0.594
119 24 1 0.41929 0.06734 0.306055 0.574
130 23 1 0.39842 0.06715 0.286338 0.554
132 22 1 0.37781 0.06678 0.267181 0.534
141 21 1 0.35745 0.06624 0.248591 0.514
152 19 2 0.31584 0.06486 0.211190 0.472
154 17 1 0.29608 0.06387 0.194001 0.452
156 16 1 0.27265 0.06289 0.173487 0.429
177 14 1 0.24811 0.06174 0.152353 0.404
185 13 1 0.22436 0.06018 0.132633 0.380
190 12 1 0.20143 0.05822 0.114311 0.355
196 11 1 0.17910 0.05588 0.097163 0.330
201 10 1 0.15675 0.05322 0.080583 0.305
245 9 1 0.13440 0.05018 0.064649 0.279
292 8 1 0.11323 0.04664 0.050506 0.254
318 7 1 0.09332 0.04259 0.038149 0.228
333 6 1 0.07474 0.03804 0.027567 0.203
402 5 1 0.05617 0.03300 0.017760 0.178
447 4 1 0.03948 0.02728 0.010191 0.153
511 3 1 0.02491 0.02093 0.004800 0.129
536 2 1 0.01224 0.01383 0.001336 0.112
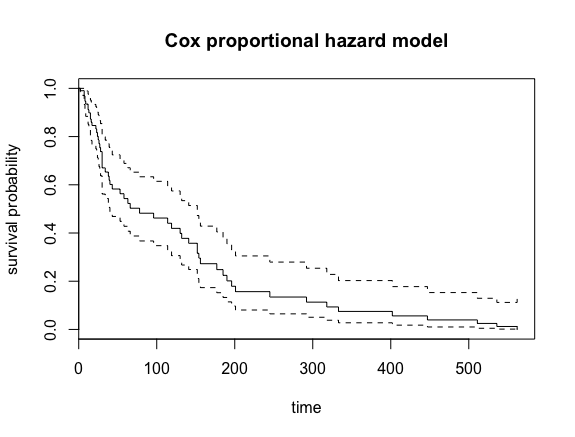
562 1 1 0.00318 0.00613 0.000073 0.139survfit関数で推定された生存曲線および信頼区間のグラフを描画してみます。
> plot(kidney.cox.fit, main="Cox proportional hazard model", xlab="time", ylab="survival probability")
Cox比例ハザード回帰分析のモデル信頼性の確認
残差分析
生存時間分析の残渣分析には、マルチンゲール残差(martingale residuals)、シェーンフィールド残差(Schoenfeld residuals)、スコア残差(score residuals)、デヴィアンス残差(deviance residuals)などいくつかの方法があります [1]。Cox比例ハザード回帰分析の残差分析はresiduals関数を用います。デフォルトでは、マルチンゲール残差が適用されますが、他の方法を指定することもできます。ここでは、デヴィアン残差のプロットも描画してみます。
ちなみに、残差分析そのものについては、このあたりが参考になりました [2]。
> scatter.smooth(residuals(kidney.cox)) # scatter.smoothは散布図を描き、Lowess平滑化 [3] による平滑化曲線を描く関数
>abline(h=0,lty=3,col=2)
> scatter.smooth(residuals(kidney.cox, type="deviance"))
> abline(h=0,lty=3,col=2)
ハザードの比例性の分析
Cox比例ハザード回帰分析は、リスクが時間の経過によらず一定という前提に基づいて行われているそうです。パッケージsurvivalには、cox.zphという比例性を分析する関数があります。
> kidney.zph<-cox.zph(kidney.cox) > kidney.zph
rho chisq p
sex 0.18839 2.60676 0.106
diseaseGN -0.01392 0.01123 0.916
diseaseAN 0.06162 0.20036 0.654
diseasePKD 0.00701 0.00438 0.947
GLOBAL NA 4.20781 0.379cox.zphで仮説が棄却されると比例ハザードの仮定が充たされていない可能性があります。今回は、すべてp>0.05なので棄却されず、比例ハザード性があると言えます。
cox.zph関数で得られた情報をplotで描画すると、シェーンフィールド残差プロットに、スプライン平滑化された曲線と標準誤差の2倍の信頼区間が示されるグラフを作成することができます。
> par(mfrow=c(1,4)) # 1列4行のレイアウトでグラフを並べる指示
> plot(kidney.zph)
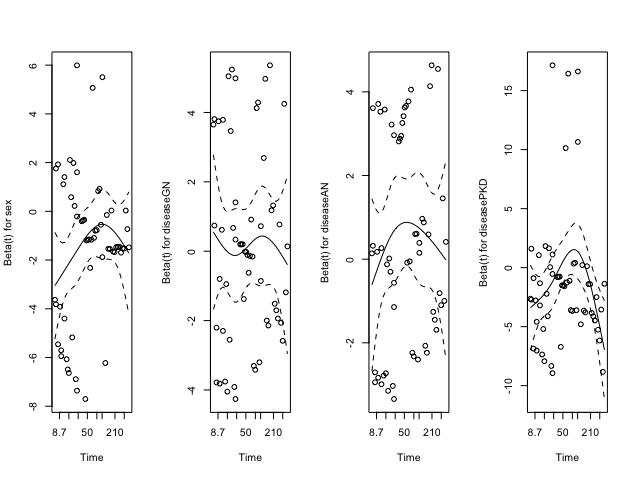
時間経過に伴うβの変化をみます。PKDが終盤にかけて意味ありげに下降していますが、許容範囲内なのでしょうか。よく分かりません。
交互作用を考慮したCox比例ハザード回帰分析
重回帰分析のように、Cox比例ハザード回帰分析でも説明変数の交互作用効果を取り入れたモデルを作ることができます。
> kidney.cox2 <- coxph(Surv(time, status) ~ (sex+disease+age)^2, data = kidney) # ^2によりそれぞれの交互作用を取り入れる
> summary(kidney.cox2)
Call:
coxph(formula = Surv(time, status) ~ (sex + disease + age)^2,
data = kidney)
n= 76, number of events= 58
coef exp(coef) se(coef) z Pr(>|z|)
sex -2.613e+00 7.330e-02 1.210e+00 -2.159 0.0308 *
diseaseGN -7.846e-01 4.563e-01 3.041e+00 -0.258 0.7964
diseaseAN -1.883e+00 1.522e-01 2.581e+00 -0.729 0.4658
diseasePKD -1.437e+01 5.761e-07 5.595e+00 -2.568 0.0102 *
age 2.107e-02 1.021e+00 7.526e-02 0.280 0.7795
sex:diseaseGN 9.954e-01 2.706e+00 1.343e+00 0.741 0.4586
sex:diseaseAN 1.301e+00 3.672e+00 1.223e+00 1.063 0.2876
sex:diseasePKD 3.710e+00 4.084e+01 1.691e+00 2.193 0.0283 *
sex:age -4.400e-03 9.956e-01 4.045e-02 -0.109 0.9134
diseaseGN:age -2.313e-02 9.771e-01 2.879e-02 -0.803 0.4217
diseaseAN:age -3.377e-03 9.966e-01 2.995e-02 -0.113 0.9102
diseasePKD:age 1.396e-01 1.150e+00 1.114e-01 1.253 0.2103
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
exp(coef) exp(-coef) lower .95 upper .95
sex 7.330e-02 1.364e+01 6.838e-03 7.858e-01
diseaseGN 4.563e-01 2.192e+00 1.177e-03 1.769e+02
diseaseAN 1.522e-01 6.571e+00 9.671e-04 2.395e+01
diseasePKD 5.761e-07 1.736e+06 9.958e-12 3.332e-02
age 1.021e+00 9.792e-01 8.812e-01 1.184e+00
sex:diseaseGN 2.706e+00 3.696e-01 1.946e-01 3.762e+01
sex:diseaseAN 3.672e+00 2.723e-01 3.340e-01 4.038e+01
sex:diseasePKD 4.084e+01 2.448e-02 1.484e+00 1.124e+03
sex:age 9.956e-01 1.004e+00 9.197e-01 1.078e+00
diseaseGN:age 9.771e-01 1.023e+00 9.235e-01 1.034e+00
diseaseAN:age 9.966e-01 1.003e+00 9.398e-01 1.057e+00
diseasePKD:age 1.150e+00 8.697e-01 9.242e-01 1.431e+00
Concordance= 0.712 (se = 0.041 )
Rsquare= 0.351 (max possible= 0.993 )
Likelihood ratio test= 32.86 on 12 df, p=0.001
Wald test = 34.17 on 12 df, p=6e-04
Score (logrank) test = 44.45 on 12 df, p=1e-05
> library(MASS) # パッケージMASSの読み込み
> stepAIC(kidney.cox2) # 交互作用も取り入れたモデルの説明変数をStepwise法にて絞り込み
Start: AIC=366.95
Surv(time, status) ~ (sex + disease + age)^2
Df AIC
- disease:age 3 362.99
- sex:age 1 364.96
366.95
- sex:disease 3 367.26
Step: AIC=362.99
Surv(time, status) ~ sex + disease + age + sex:disease + sex:age
Df AIC
- sex:age 1 361.36
362.99
- sex:disease 3 368.67
Step: AIC=361.36
Surv(time, status) ~ sex + disease + age + sex:disease
Df AIC
- age 1 359.54
361.36
- sex:disease 3 368.16
Step: AIC=359.54
Surv(time, status) ~ sex + disease + sex:disease
Df AIC
359.54
- sex:disease 3 366.24
Call:
coxph(formula = Surv(time, status) ~ sex + disease + sex:disease,
data = kidney)
coef exp(coef) se(coef) z p
sex -2.5252779 0.0800361 0.5659622 -4.462 8.12e-06
diseaseGN -1.3415792 0.2614325 1.3037724 -1.029 0.303481
diseaseAN -1.4180768 0.2421793 1.4840553 -0.956 0.339304
diseasePKD -7.3819153 0.0006224 1.9493785 -3.787 0.000153
sex:diseaseGN 0.8312922 2.2962840 0.7604435 1.093 0.274320
sex:diseaseAN 1.0754002 2.9311656 0.8156353 1.318 0.187342
sex:diseasePKD 4.2144726 67.6584758 1.1501090 3.664 0.000248
Likelihood ratio test=30.26 on 7 df, p=8.496e-05
n= 76, number of events= 58 ステップワイズ法の結果を見ると、性別とPKD(多発性嚢胞腎)の間に交互作用があり、モデルの有意な説明因子になっていることが分かります。
最後に、交互作用がどうなっているのか性別と疾患で層別化した分析を行って、確認してみます。
> survfit(Surv(time,status) ~sex+disease, data=kidney)
Call: survfit(formula = Surv(time, status) ~ sex + disease, data = kidney)
n events median 0.95LCL 0.95UCL
sex=1, disease=Other 6 6 19 12 NA
sex=1, disease=GN 6 6 12 7 NA
sex=1, disease=AN 4 3 17 12 NA
sex=1, disease=PKD 4 3 152 63 NA
sex=2, disease=Other 20 14 185 119 NA
sex=2, disease=GN 12 8 132 30 NA
sex=2, disease=AN 20 15 58 39 NA
sex=2, disease=PKD 4 3 78 30 NA多発性嚢胞腎の場合、男性より女性の方が倍近く早く感染症を起こしやすいことが確認できます。交互作用の確認は大切なんですね。
[1] mitsurukikkawa – 生存時間分析(survival analysis) [2] 統計WEB – 27-3. 予測値と残差 [3] MathWorks – Lowess 平滑化Sponsored Links


